library(splancs)
library(sp)
library(maptools)
path <- “E:/workspace/IDE/Rworkspace/核密度/”
uscp <- read.csv(paste(path,”2014_us_cities.csv”,sep = “”),header = T)
#################################################################
#
# 纯空间位置无属性加权的核密度分析
#
################################################################
#
#生成一个空间的矩阵,行数和uscp一样,列数为2,用来灌装点数据
#
sp_point <- matrix(NA, nrow=length(uscp[,1]),ncol=2)
#
#将经纬度灌装到矩阵里面,并且设置列名为X/Y
#
sp_point[,1] <- uscp$lon
sp_point[,2] <- uscp$lat
colnames(sp_point) <- c(“X”,”Y”)
#
#生成一个矩形,R语言生成矩形的顺序是从右下角开始,逆时针构建四个顶点的如下:
# 3 2
# 4 1
#
poly <- as.points(c(min(sp_point[,1]),max(sp_point[,1]),
max(sp_point[,1]),min(sp_point[,1])),
c(max(sp_point[,2]),max(sp_point[,2]),
min(sp_point[,2]),min(sp_point[,2])))
#
#构建以个Point对象(sp包里面)的,并且设置空间参考为wgs84
#
sp_points <- SpatialPoints(coords=sp_point, proj4string=CRS(“+proj=longlat +datum=WGS84”))
#
#下面这一段是构建核密度栅格图的各种参数
#核密度计算结果,最后可以体现为一个栅格,栅格的基本参数有如下三个:
#################################################################
# x/y min :左下角坐标
# cellsize : 栅格单元的大小。
# dim :总的栅格行列数(这个参数有上两个,就可以算出来了,但是R语言保留了这个
# 参数,也就是说,你可以自由控制这个栅格的大小和范围,更为灵活。
#################################################################
#
cellsize = c(0.1,0.1)
xymin = c(min(sp_point[,1]),min(sp_point[,2]))
xymax = c(max(sp_point[,1]),max(sp_point[,2]))
cdim = (xymax – xymin)/cellsize
#
# 构建一个栅格,参数用上面那些参数
#
grdx <- GridTopology(xymin,cellsize=cellsize,cells.dim=cdim)
#
# 进行核密度计算,需要的参数有点、范围、带宽、生成的结果栅格
# 生成的结果是一个巨大的数字型向量。
#
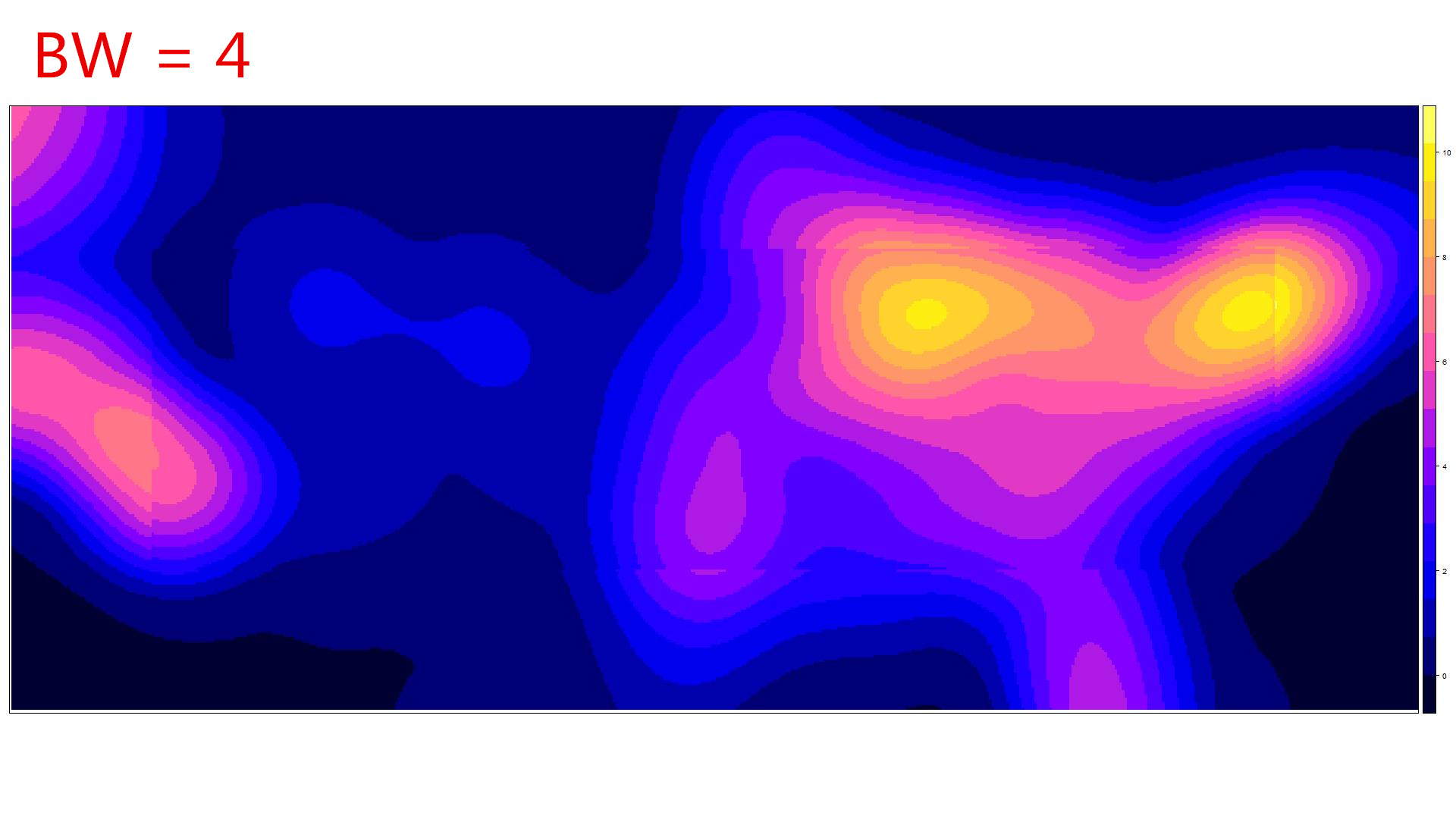
kernel1 <- spkernel2d(sp_points, poly=poly, h0=4, grd=grdx)
#
# 把生成的核密度结果变成一个数据框
# 然后把这个数据框和构建的空栅格挂接起来,变成一个Grid
#
df <-data.frame(kernel1 = kernel1)
SG <- SpatialGridDataFrame(grdx, data=df)
#
# 调用sp包里面的spplot绘图,当然也可以直接保存成图片。
#
spplot(SG)
#
# 下面同时画出四个带宽的核密度图,并且用同一个渲染尺度进行渲染
#
kernel1 <- spkernel2d(sp_points, poly=poly, h0=0.5, grd=grdx)
kernel2 <- spkernel2d(sp_points, poly=poly, h0=1, grd=grdx)
kernel3 <- spkernel2d(sp_points, poly=poly, h0=2, grd=grdx)
kernel4 <- spkernel2d(sp_points, poly=poly, h0=4, grd=grdx)
df <-data.frame(kernel4 = kernel4,kernel3 = kernel3,
kernel2 = kernel2,kernel1 = kernel1)
jpeg(“e:/kall.jpg”,width = 1920,height = 1080)
SG <- SpatialGridDataFrame(grdx, data=df)
spplot(SG)
dev.off() |